Científicos del Instituto Tecnológico de Massachusetts (MIT) han publicado recientemente un potente modelo de inteligencia artificial de código abierto llamado Boltz-1. Esta innovación promete acelerar significativamente la investigación biomédica y el desarrollo de fármacos.
Boltz-1 es el primer modelo completamente de código abierto que alcanza el mismo nivel de sofisticación que AlphaFold3 de Google DeepMind en la predicción de estructuras biomoleculares. El equipo de desarrollo, perteneciente a la Clínica de Aprendizaje Automático Jameel del MIT, está liderado principalmente por los estudiantes de posgrado Jeremy Walwen y Gabriel Corso, en colaboración con el investigador del MIT Saro Passaro y los profesores de ingeniería eléctrica e informática Regina Barzilay y Tommy Jacques.
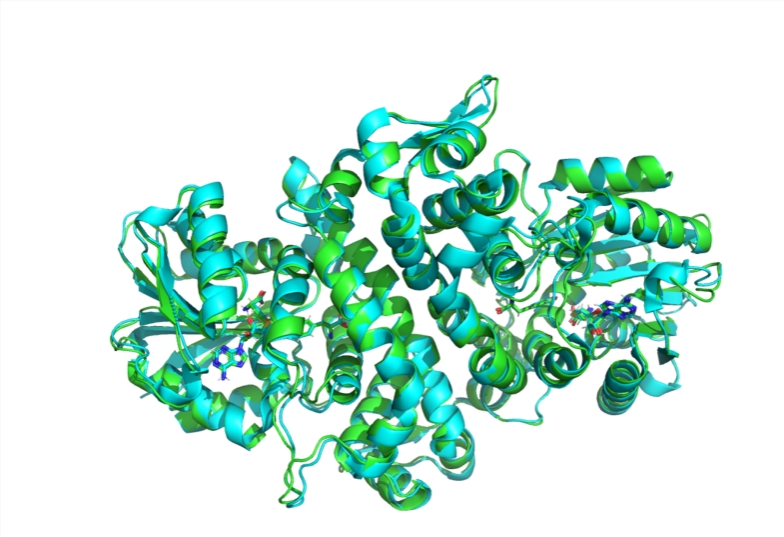
En una presentación el 5 de diciembre, Walwen y Corso declararon que su objetivo final es fomentar la colaboración global, acelerar los descubrimientos científicos y proporcionar una plataforma sólida para avanzar en el modelado biomolecular. Corso mencionó: "Esperamos que esto sea un punto de partida para la comunidad", y enfatizó que el nombre "Boltz-1" en lugar de "Boltz" pretende alentar la participación de la comunidad.
Las proteínas juegan un papel crucial en casi todos los procesos biológicos, y su forma está estrechamente relacionada con su función. Por lo tanto, comprender la estructura de las proteínas es fundamental para diseñar nuevos fármacos o proteínas nuevas con funciones específicas. Debido a la complejidad extrema del proceso de plegamiento de las largas cadenas de aminoácidos de las proteínas en estructuras tridimensionales, la predicción precisa de su estructura ha sido un desafío importante en el ámbito científico.
AlphaFold2 de DeepMind, mediante el aprendizaje automático, predice rápidamente las estructuras de proteínas 3D con una precisión tan alta que resulta difícil de distinguir para los científicos experimentales. AlphaFold3, una mejora de AlphaFold2, utiliza un modelo de IA generativo, pero su falta de código abierto ha sido criticada por la comunidad científica. Por lo tanto, el equipo de investigación del MIT se propuso desarrollar Boltz-1, siguiendo los principios básicos de AlphaFold3 y mejorándolos para aumentar la precisión y la eficiencia de la predicción.
El equipo de investigación dedicó cuatro meses a realizar múltiples experimentos, superando problemas de ambigüedad y heterogeneidad encontrados en el banco de datos de proteínas. Finalmente, sus experimentos demostraron que Boltz-1 alcanza la misma precisión que AlphaFold3 en la predicción de estructuras biomoleculares complejas.
Los investigadores planean continuar mejorando el rendimiento de Boltz-1 y reducir el tiempo de predicción. También invitan a otros investigadores a probar Boltz-1 en GitHub e intercambiar ideas con otros usuarios a través de un canal de Slack. El equipo espera que Boltz-1 fomente una colaboración más amplia e impulse aplicaciones creativas de la comunidad.
Proyecto: https://jclinic.mit.edu/democratizing-science-boltz-1/
Puntos clave:
🌍 Boltz-1 es el primer modelo de predicción de estructuras biomoleculares completamente de código abierto que alcanza el mismo rendimiento que AlphaFold3.
🧬 El desarrollo de este modelo tiene como objetivo fomentar la colaboración global y promover la investigación biomédica y el desarrollo de fármacos.
🔬 El equipo del MIT espera que Boltz-1 simplifique la predicción de estructuras de proteínas, permitiendo que más investigadores puedan utilizar esta poderosa herramienta.

