Une équipe de recherche de Google en collaboration avec des chercheurs de l'Université de Californie à Santa Cruz a lancé DeepSomatic, un modèle d'intelligence artificielle innovant conçu pour identifier les variations génétiques dans les cellules cancéreuses. Dans une étude menée en coopération avec le Children's Hospital, DeepSomatic a réussi à identifier 10 variantes de leucémie infantile que d'autres outils n'avaient pas détectées.
DeepSomatic utilise un appelateur de petites variations spécifique au génome du cancer, compatible avec les lectures courtes d'Illumina, les lectures longues HiFi de PacBio et les lectures longues de Oxford Nanopore. Cette méthode est une extension de DeepVariant, capable de détecter les variations mononucléotidiques (SNV) et les insertions-délétions (indels), soutenant les workflows tumorales-normaux et tumorales seules, y compris le modèle de fixation au formol et de paraffine (FFPE).
Le fonctionnement de DeepSomatic consiste à convertir les lectures alignées en tenseurs ressemblant à des images, qui codent les situations de superposition, la qualité de base et le contexte d'alignement. Grâce au réseau neuronal convolutif, le modèle classe les sites candidats comme étant des variations somatiques ou non, générant finalement des fichiers VCF ou gVCF. Ce design permet à DeepSomatic d'être très adaptable aux plateformes techniques, car ces tenseurs résument les modèles locaux de haplotype et les erreurs entre les différentes technologies.
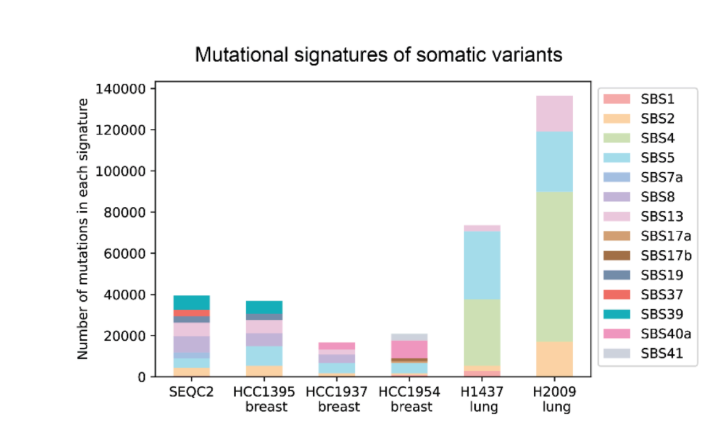
En ce qui concerne les jeux de données et les tests de référence, DeepSomatic utilise le jeu de données CASTLE (évaluation standard des lectures longues des cancers) pour l'entraînement et l'évaluation. Ce jeu de données comprend 6 paires de lignées cellulaires tumorales et normales, séquencées à l'aide d'Illumina, PacBio HiFi et Oxford Nanopore. L'équipe de recherche a publié un ensemble de référence et a donné l'accès afin que d'autres chercheurs puissent le réutiliser, combler ainsi le vide concernant les ressources d'entraînement et de test multi-technologies.
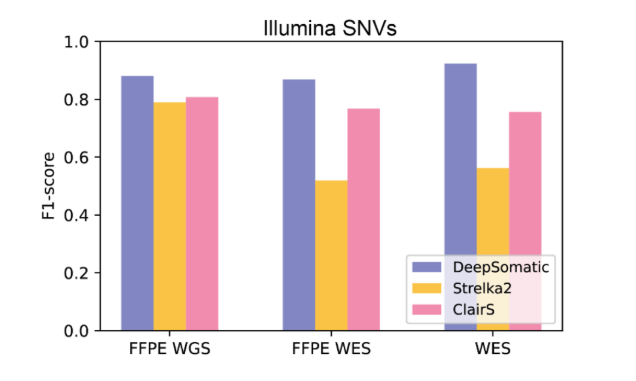
Les résultats montrent que DeepSomatic dépasse les méthodes largement utilisées actuellement lors de la détection des variations mononucléotidiques et des insertions-délétions. Par exemple, concernant les insertions-délétions séquencées par Illumina, DeepSomatic atteint un score F1 d'environ 90 %, tandis que les autres méthodes ne dépassent pas 80 %. Sur les séquences PacBio, le score F1 de DeepSomatic dépasse 80 %. L'équipe de recherche a également rapporté la découverte de 329 011 variations somatiques, confirmant ainsi la forte capacité de DeepSomatic dans la détection des insertions-délétions.
Recherche : https://research.google/blog/using-ai-to-identify-genetic-variants-in-tumors-with-deepsomatic/
Points clés :
🌟 DeepSomatic est capable de reconnaître diverses variations génétiques dans les cellules cancéreuses, couvrant plusieurs plateformes de séquençage.
🔍 Le modèle utilise un réseau neuronal convolutif pour transformer les informations de lecture en tenseurs ressemblant à des images, assurant ainsi précision et cohérence.
📊 Dans les tests de référence, la précision de détection de DeepSomatic dépasse clairement les méthodes actuelles, notamment dans la détection des insertions-délétions.
