El equipo de investigación de Google en colaboración con investigadores de la Universidad de California, Santa Cruz, lanzó DeepSomatic, un modelo innovador de inteligencia artificial diseñado para identificar variantes genéticas en células cancerosas. En una investigación conjunta con el Centro Médico Infantil, DeepSomatic descubrió con éxito 10 mutaciones en células de leucemia infantil que no pudieron ser identificadas por otras herramientas.
DeepSomatic utiliza un llamador de variaciones pequeñas específico para genomas de cáncer, compatible con lecturas cortas de Illumina, lecturas largas de PacBio HiFi y lecturas largas de Oxford Nanopore. Este método se basa en DeepVariant y es capaz de detectar variantes de nucleótido único (SNV) y pequeñas inserciones y delecciones (indels), soportando flujos de trabajo tumor-normal y tumor-único, incluyendo modelos de fijación con formalina y embebidos en parafina (FFPE).
El funcionamiento de DeepSomatic consiste en convertir las lecturas alineadas en tensores similares a imágenes, los cuales codifican información sobre la superposición, la calidad base y el contexto de alineación. A través de redes neuronales convolucionales, el modelo clasifica los sitios candidatos como variantes somáticas o no variantes, generando finalmente archivos VCF o gVCF. Este diseño hace que DeepSomatic sea altamente adaptable a diversas plataformas tecnológicas, ya que estos tensores resumen los patrones locales de haplotipos y errores entre distintas tecnologías.
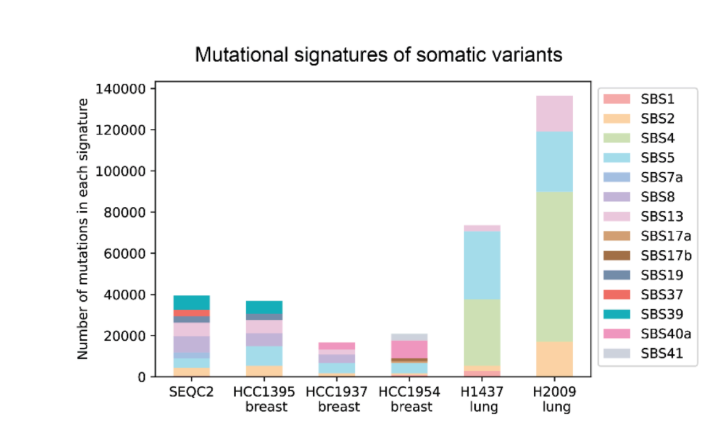
En cuanto a conjuntos de datos y pruebas de referencia, DeepSomatic se entrena y evalúa utilizando el conjunto de datos CASTLE (Evaluación Estándar de Lecturas Largas para Cáncer). Este conjunto contiene 6 pares de líneas celulares tumorales y normales, secuenciadas completamente con Illumina, PacBio HiFi y Oxford Nanopore. El equipo de investigación publicó conjuntos de referencia y acceso para que otros investigadores los reutilicen, lo cual cubre la brecha en recursos para entrenamiento y prueba somático multitecnológico.
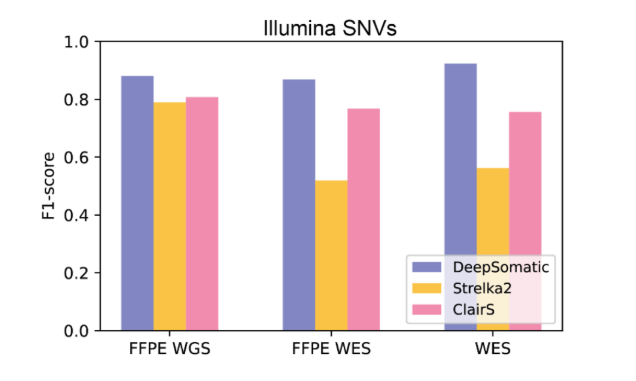
Los resultados muestran que DeepSomatic supera a los métodos ampliamente utilizados actualmente en la detección de variantes de nucleótido único y pequeñas inserciones y delecciones. Por ejemplo, en la detección de inserciones y delecciones en secuenciación de Illumina, DeepSomatic alcanzó aproximadamente un 90% de puntuación F1, mientras que otros métodos alcanzaron solo un 80%. En la secuenciación de PacBio, la puntuación F1 de DeepSomatic superó el 80%. El equipo de investigación también informó del descubrimiento de 329.011 variantes somáticas, validando así la capacidad del DeepSomatic en la detección de inserciones y delecciones.
Investigación: https://research.google/blog/using-ai-to-identify-genetic-variants-in-tumors-with-deepsomatic/
Resumen:
🌟 DeepSomatic es capaz de identificar varias variantes genéticas en células cancerosas, abarcando múltiples plataformas de secuenciación.
🔍 El modelo utiliza una red neuronal convolucional para convertir la información de lectura en tensores visuales, asegurando precisión y consistencia.
📊 En pruebas de referencia, la precisión de detección de DeepSomatic es significativamente superior a los métodos actuales, especialmente en la detección de inserciones y delecciones.