Das Forschungsteam von Google und Forscher der University of California, Santa Cruz haben DeepSomatic vorgestellt, ein innovatives KI-Modell, das darauf abzielt, Gen变异 in Krebszellen zu identifizieren. In einer Zusammenarbeit mit dem Children's Hospital hat DeepSomatic 10 kindliche Leukämiezell-Variationen entdeckt, die andere Tools nicht erkennen konnten.
DeepSomatic verwendet einen kleinen Varianten-Caller, der speziell für Krebsgenome entwickelt wurde, und ist kompatibel mit Illumina-Kurzlesungen, PacBio HiFi-Langlesungen und Oxford Nanopore-Langlesungen. Dieser Ansatz baut auf DeepVariant auf und kann Einzelnukleotid-Varianten (SNV) und kleine Insertionen und Deletionen (indels) detektieren. Er unterstützt Workflows für Tumor-Normal und Tumor-Einzelfall, einschließlich des Formalin-fixierten Paraffin-eingebetteten (FFPE)-Modells.
Die Funktionsweise von DeepSomatic besteht darin, alignierte Lesungen in bilddatenartige Tensoren umzuwandeln, die Informationen über Stapelung, Grundqualität und Alignment-Kontext codieren. Durch Convolutional Neural Networks klassifiziert das Modell Kandidatennstellen als somatische Variante oder Nicht-Variante und generiert schließlich VCF- oder gVCF-Dateien. Diese Design ermöglicht DeepSomatic eine hohe Anpassungsfähigkeit an verschiedene Technologien, da diese Tensoren lokale Haplotypen und Fehlermuster zwischen verschiedenen Technologien zusammenfassen.
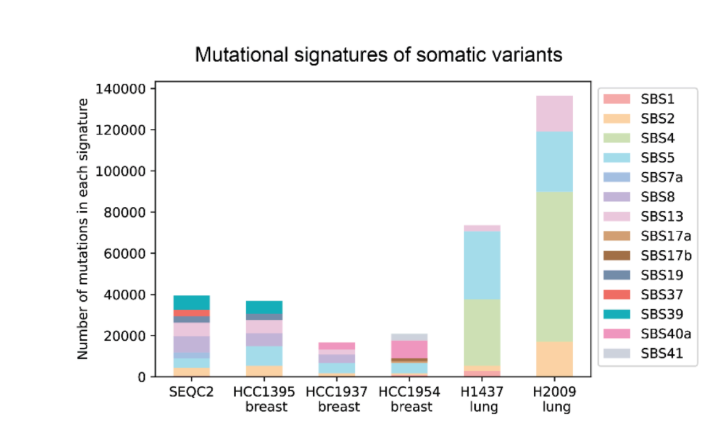
In Bezug auf Datensätze und Benchmarks wird DeepSomatic mit dem CASTLE-Datensatz (Cancer Standard Long Read Evaluation) trainiert und bewertet. Der Datensatz enthält 6 Paare von Tumor- und Normalzelllinien, die mit Illumina, PacBio HiFi und Oxford Nanopore vollständig genomsequenziert wurden. Das Forschungsteam hat Benchmarks und Zugriffsrechte veröffentlicht, um anderen Forschern die Wiederverwendung zu ermöglichen, was eine Lücke bei Ressourcen für multitechnische somatische Trainings- und Testdaten schließt.
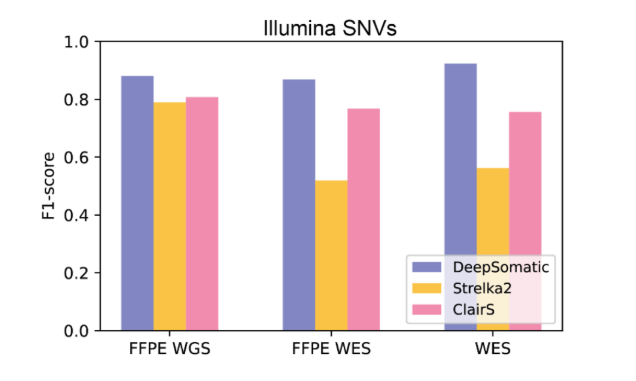
Die Ergebnisse zeigen, dass DeepSomatic sowohl bei der Erkennung von Einzelnukleotid-Varianten als auch bei kleinen Insertionen und Deletionen besser abschneidet als derzeit weit verbreitete Methoden. Zum Beispiel erreichte DeepSomatic bei Insertionen und Deletionen aus Illumina-Sequenzierungen einen F1-Score von etwa 90 %, während andere Methoden nur 80 % erreichten; bei PacBio-Sequenzierungen überstieg der F1-Score von DeepSomatic 80 %. Das Forschungsteam berichtete außerdem von der Entdeckung von 329.011 somatischen Varianten, was die starke Fähigkeit von DeepSomatic bei der Erkennung von Insertionen und Deletionen weiter bestätigte.
Forschung: https://research.google/blog/using-ai-to-identify-genetic-variants-in-tumors-with-deepsomatic/
Wichtige Punkte:
🌟 DeepSomatic kann verschiedene genetische Varianten in Krebszellen identifizieren und unterstützt verschiedene Sequenzierungsplattformen.
🔍 Das Modell verwendet ein convolutionales neuronales Netzwerk, um Lesedaten in bilddatenartige Tensoren umzuwandeln, um Genauigkeit und Konsistenz sicherzustellen.
📊 Im Benchmark-Test schneidet DeepSomatic deutlich besser ab als bestehende Mainstream-Methoden, insbesondere bei der Erkennung von Insertionen und Deletionen.







